
BsaI ຂໍ້ຈໍາກັດ Endonuclease
ລາຍລະອຽດ
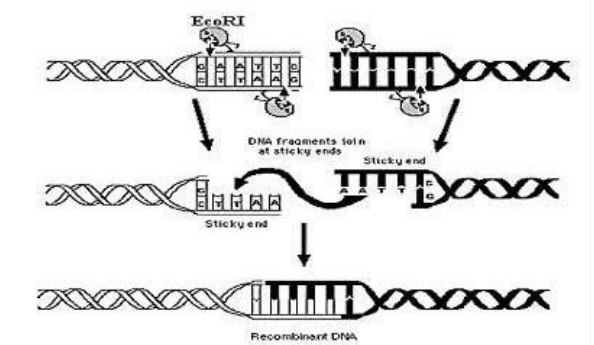
enzyme ການຈໍາກັດ BsaI ແມ່ນ endonuclease ປະເພດ II ທີ່ຈໍາກັດໄດ້ມາຈາກທາດໂປຼຕີນຈາກ recombinant ເຂົ້າລະຫັດໂດຍ gene BspQI ໃນ Bacillus sphaericus ສະແດງອອກໂດຍ E.coli.ການຈໍາກັດ endonucleases ຖືກນໍາໃຊ້ຢ່າງກວ້າງຂວາງໃນການສ້າງແຜນທີ່ gene, cloning ແລະຂົງເຂດອື່ນໆ.ເອນໄຊນີ້ຕັດ DNA ຢ່າງໄວວາສໍາລັບ gene linearization Bsa I ທີ່ມີປະສິດຕິພາບ, ການຈໍາກັດການຈໍາກັດ endonuclease IIs, ແມ່ນມາຈາກສາຍພັນ E. coli ທີ່ປະສົມພັນທີ່ບັນຈຸພັນທຸກໍາ BsaI ທີ່ຖືກໂຄນແລະດັດແປງຈາກ Bacillus stearothermophilus.ເຊິ່ງມີຄວາມສະເພາະດຽວກັນກັບ enzymes ພື້ນເມືອງແລະກິດຈະກໍາດາວຫຼຸດລົງ.ລຳດັບການຮັບຮູ້ ແລະສະຖານທີ່ແຕກແຍກຂອງມັນມີດັ່ງນີ້:
5'······ GGTTC(N)·········3'
3.
ໂຄງສ້າງທາງເຄມີ
ຄຸນສົມບັດຂອງຜະລິດຕະພັນ
• endonuclease ຈໍາກັດມີກິດຈະກໍາ enzymatic ສູງແລະສໍາເລັດຕິກິຣິຍາການຍ່ອຍອາຫານຢ່າງໄວວາ.
•ກິດຈະກໍາ endonuclease ຕ່ໍາທີ່ບໍ່ສະເພາະເພື່ອຮັບປະກັນການຕັດ "scalpel" ທີ່ຊັດເຈນ.
•ບໍ່ມີລະບົບ BSA, ຫຼຸດຜ່ອນມົນລະພິດຂອງແຫຼ່ງຄວາມຮ້ອນ.
ຂໍ້ມູນຈໍາເພາະ
| ລາຍການທົດສອບ | ຂໍ້ມູນຈໍາເພາະ |
| ກິດຈະກໍາຂອງເອນໄຊ | 20 U/µL |
| ຄວາມບໍລິສຸດ(SDS-PAGE) | ≥95% |
| Rnase | ບໍ່ກວດພົບ |
| ກິດຈະກໍາ DNase ທີ່ບໍ່ສະເພາະ | ບໍ່ກວດພົບ |
| ກິດຈະກຳຕິດດາວ | ບໍ່ກວດພົບ |
| ການຕັດສາຍພັນແລະການຕັດ | ຫຼັງຈາກການຍ່ອຍອາຫານ, ຊິ້ນ DNA ສາມາດຖືກຜູກມັດ.ແລະຊິ້ນສ່ວນທີ່ຜູກມັດເຫຼົ່ານີ້ສາມາດຖືກຕັດດ້ວຍ Bsal. |
| Endotoxins (ການທົດສອບ LAL) | ≤10 EU/mg |
| E.Coli DNA | ≤1 ສຳເນົາ/2U |
ການຄວບຄຸມຄຸນນະພາບ
ຄວາມບໍລິສຸດ ≥ 95%, ບໍ່ມີ DNase, ກິດຈະກໍາ RNase, residue DNA ເຈົ້າພາບ ≤ 100pg/mg, ທາດໂປຼຕີນຈາກ host residue ≤ -50ppm, endotoxin residue ≤ 10EU/mg, ບໍ່ມີກິດຈະກໍາ protease, ເປັນຫມັນ, ບໍ່ມີ mycoplasma.
ຄໍາຮ້ອງສະຫມັກ
ຍ່ອຍ plasmid ເພື່ອກະກຽມຊິ້ນສ່ວນ DNA ທີ່ເປັນເສັ້ນຢູ່ໃນຕອນທ້າຍຂອງ Poly (A/T/C/G);
Linearize plasmid template ກ່ອນ in vitro transcription;
ຍ່ອຍຂໍ້ຈໍາກັດ;
ນິຍາມຫົວໜ່ວຍ
ຫນຶ່ງຫນ່ວຍແມ່ນຖືກກໍານົດເປັນຈໍານວນເອນໄຊທີ່ຕ້ອງການເພື່ອຍ່ອຍ 1 µg ຂອງ DNA ຄວບຄຸມພາຍໃນໃນ 1 ຊົ່ວໂມງຢູ່ທີ່ 37 ° C ໃນປະລິມານຕິກິຣິຍາທັງຫມົດ 50 µL.
ການຂົນສົ່ງແລະການເກັບຮັກສາ
ການຂົນສົ່ງ:ຈັດສົ່ງພາຍໃຕ້ -15 ° C
ການເກັບຮັກສາ:ເກັບຮັກສາໄວ້ທີ່ -25 ~ -15 ອົງສາ C
ການທົດສອບຊີວິດທີ່ແນະນໍາໃຫ້:1 ປີ















